CRISPR反式切割的发现历程,诠释了基础研究的不可预测性及其转化潜力。从最初被视为实验干扰因素,到成为诊断技术的核心,这一"意外超能力"的开发利用,体现了科学探索中保持开放思维的重要性。


2015年至2018年,研究人员意外发现CRISPR系统的一个独特现象:某些Cas蛋白识别并切割目标核酸序列后,实验中的单链核酸报告分子也被意外切割。这个看似“失误”的现象,催生了一项可能改变医学诊断格局的新发现——Cas蛋白的反式切割特性。如今,基于这种“意外超能力”开发的检测方法,已在全球范围内广泛应用于新冠病毒、HPV等病原体的快速检测。
CRISPR反式切割:一体两面的剪刀
研究人员发现,某些Cas蛋白在向导RNA(gRNA)引导激活后,会展现出对单链核酸分子的非特异性切割能力。这一特性被称为反式切割,它如同一把“双刃剑”,在不同应用场景中展现出截然不同的影响。这种反式切割机制就像一把“失控的剪刀”:当Cas蛋白被激活后,它不仅会精准切割目标序列,还会不加区分地切割周围的单链DNA或RNA分子。
在基因编辑领域,这种非特异性切割能力可能带来严重副作用:非特异性切割可能破坏细胞内与转录和翻译相关的核酸分子,导致严重细胞毒性,甚至引发细胞功能障碍和死亡。
然而,在核酸检测领域,这一特性却展现出巨大优势:在检测目标病毒(如新冠病毒)时,Cas蛋白的非特异性切割能够持续降解报告分子,产生“雪球效应”的信号放大效果,大幅提升检测灵敏度。与传统的顺式切割系统相比,基于反式切割的检测方法不仅操作更简便、检测灵敏度更高,还能适应家庭或野外等复杂环境下的即时检测需求。
目前已知的CRISPR/Cas系统中,Cas12、 13和14系统是被广泛证实拥有反式切割能力的生物效应器。
分子机制的奥秘:蛋白结构特性
Cas蛋白切割活性的差异源于其结构的根本不同。Cas9在切割DNA后保持结构稳定,而具有反式切割特性的Cas蛋白在识别目标后会经历显著的构象变化。
Cas12包含RuvC和Nuc两个核酸酶结构域。其中,RuvC主要负责结合和切割DNA,而Nuc则用于辅助协调RuvC的功能。当目标DNA与向导RNA配对后,Cas12a的REC结构域(负责识别DNA的部分)像开关一样移动,触发整个蛋白的构象变化。这种构象变化使得原本隐藏的RuvC结构域(核酸酶活性中心)暴露出来,进而切断单链核酸。
与Cas12不同,Cas13利用两个HEPN结构域切割RNA。当目标RNA与Cas13a-crRNA复合体结合时,这两个HEPN结构域会相互靠近,形成完整的催化中心。
Cas14a(Cas12f)相比于其他CRISPR蛋白小得多,当Cas14a的crRNA+tracrRNA复合物(gRNA)识别到目标ssDNA时,蛋白构象会发生变化,并类似于Cas12通过RuvC结构对单核酸链进行切割。
基于反式切割原理,科学家开发了多种革命性检测平台:
SHERLOCK利用Cas13a检测RNA病毒(如寨卡病毒),通过“切完目标序列后乱切其他RNA”的方式放大信号。虽然灵敏度极高,但操作步骤较为复杂。升级版SHERLOCK v2能够同时检测多种病毒。
DETECTR利用Cas12a直接检测DNA病毒(如HPV),省去了RNA转换步骤,但受PAM序列的限制。
HOLMES v1版采用PCR结合Cas12a,整个流程包括了45 min的 PCR循环和15 min的荧光检测。1小时内即可出结果。
HOLMES v2版改用耐高温Cas12b,搭配LAMP恒温扩增技术,单管一步完成检测,设备要求低且具备防污染能力,但反应的灵敏度较常规两步法有所降低。
在2020年新冠疫情中,改良版STOPCovid v2方法利用Cas13检测新冠病毒RNA,仅需30分钟,成本不到10美元。该系统还创新性地采用侧向流动试纸条读取结果,使检测过程如同使用验孕试纸般简便。
这些技术让核酸检测更便携(可野外使用)、更精准(识别单碱基差异),但仍有假阳性和成本问题待解决。
表1 具有反式切割能力的Cas蛋白概览
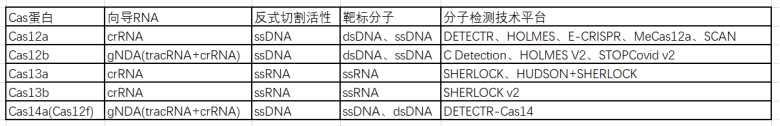
表2 中国已上市CRISPR分子检测平台试剂盒
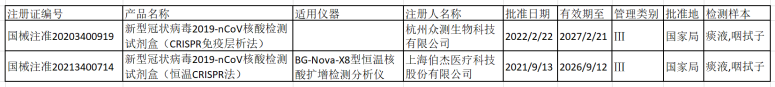
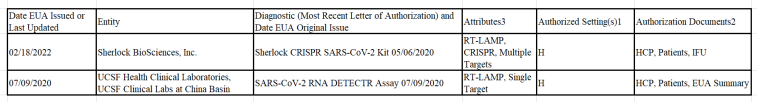
表3 美国EUA CRISPR分子检测平台产品
CRISPR反式切割的发现历程,诠释了基础研究的不可预测性及其转化潜力。从最初被视为实验干扰因素,到成为诊断技术的核心,这一"意外超能力"的开发利用,体现了科学探索中保持开放思维的重要性。
反式切割的发现不仅革新了分子诊断,更为我们展示了生物技术的无限可能——在肉眼不可见的分子世界里,这些微小的蛋白质机器正在重新定义医学检测的边界,为人类健康带来新的希望。
参考文献:
[1]Karvelis, T., et al. PAM recognition by miniature CRISPR-Cas12f nucleases triggers programmable double-stranded DNA target cleavage [J]. Nucleic Acids Res, 2020, 48(9): 5016-5023.
[2]GOOTENBERG J S, ABUDAYYEH O O, LEE J W, et al. Nucleic acid detection with CRISPR-Cas13a/C2c2[J]. Science, 2017, 356(6336): 438-442. DOI:10.1126/science.aam9321.
[3]LI S Y, CHENG Q X, WANG J M, et al. CRISPR-Cas12a-assisted nucleic acid detection[J]. Cell Discovery, 2018, 4: 20. DOI:10.1038/ s41421-018-0028-z.
[4]Stella, S., Alcón, P. & Montoya, G. Structure of the Cpf1 endonuclease R-loop complex after target DNA cleavage. Nature 546, 559–563 (2017). https://doi.org/10.1038/nature22398




发表评论 取消回复